- Profesor: pharbiois science for life
pharbiois.milaulas.com
-
Bienvenido a su nuevo sitio en Mil Aulas
¡Ya puede empezar a crear cursos!
Moodle es un sistema de gestión de aprendizaje de código abierto que permite a los educadores crear y administrar cursos en línea. Ofrece una variedad de herramientas y recursos para organizar el material del curso, crear actividades y cuestionarios, alojar discusiones en línea y hacer un seguimiento del progreso de los estudiantes.
La plataforma es altamente personalizable y puede adaptarse a las necesidades específicas de cada institución o curso. Esto significa que los educadores pueden crear cursos que se ajusten a sus objetivos y metodologías, y personalizarlos para el aprendizaje de cada estudiante. Moodle también admite tanto entornos de aprendizaje síncronos como asíncronos, lo que permite a los educadores organizar eventos en vivo y proporcionar recursos para el aprendizaje a su propio ritmo.
Moodle es ampliamente utilizado en instituciones educativas de todo el mundo, desde escuelas primarias hasta universidades. También es popular en programas de formación profesional y empresarial. Además, la plataforma es accesible para usuarios de diferentes habilidades y necesidades, lo que la hace inclusiva para todos los estudiantes.
La comunidad de Moodle es activa y diversa, compuesta por educadores, desarrolladores y usuarios de todo el mundo. La comunidad contribuye al desarrollo y mejora continua de Moodle, y proporciona soporte, recursos y documentación para los usuarios. La plataforma también ofrece una gran cantidad de recursos y herramientas para mejorar la experiencia de los usuarios y fomentar la colaboración y el aprendizaje interactivo.
Enlaces de interés:
- Documentación oficial de Moodle
- Foros de la comunidad Moodle
- Historial de versiones de Moodle (En inglés)
- El servicio de alojamiento gratuito de Moodle en Mil Aulas
(Puede editar o eliminar este texto)
Cursos disponibles
El Diplomado está diseñado para quienes buscan aprender a diseñar, analizar e interpretar datos en investigaciones biomédicas y farmacéuticas. A lo largo del diplomado se abordarán temas desde el diseño de un protocolo experimental, el cálculo del tamaño de muestra y la organización de bases de datos, hasta la selección adecuada de pruebas estadísticas según el tipo de estudio. El Diplomado es teórico-práctico, con ejercicios reales usando Excel, SPSS y GraphPad (en versiones académicas de prueba), y se imparte de manera virtual asincrónica, con posibilidad de sesiones en vivo para resolver dudas. El temario está estructurado en tres niveles: básico, intermedio y avanzado, e incluye temas como estadística descriptiva, pruebas de normalidad, chi cuadrada, ANOVA, regresión, curvas ROC, modelos lineales, validación de instrumentos y más. Se recomienda tener conocimientos básicos en investigación y manejo de hojas de cálculo. Esta formación es útil para estudiantes de posgrado, investigadores, profesionales del área de la salud y personal de la industria farmacéutica que deseen mejorar sus capacidades en análisis de datos y toma de decisiones basada en evidencia.
- Profesor: Cindy Bandala
El Diplomado está diseñado para quienes buscan aprender a diseñar, analizar e interpretar datos en investigaciones biomédicas y farmacéuticas. A lo largo del diplomado se abordarán temas desde el diseño de un protocolo experimental, el cálculo del tamaño de muestra y la organización de bases de datos, hasta la selección adecuada de pruebas estadísticas según el tipo de estudio. El Diplomado es teórico-práctico, con ejercicios reales usando Excel, SPSS y GraphPad (en versiones académicas de prueba), y se imparte de manera virtual asincrónica, con posibilidad de sesiones en vivo para resolver dudas. El temario está estructurado en tres niveles: básico, intermedio y avanzado, e incluye temas como estadística descriptiva, pruebas de normalidad, chi cuadrada, ANOVA, regresión, curvas ROC, modelos lineales, validación de instrumentos y más. Se recomienda tener conocimientos básicos en investigación y manejo de hojas de cálculo. Esta formación es útil para estudiantes de posgrado, investigadores, profesionales del área de la salud y personal de la industria farmacéutica que deseen mejorar sus capacidades en análisis de datos y toma de decisiones basada en evidencia.
- Profesor: Cindy Bandala

El Diplomado tiene como objetivo capacitar a los participantes en el uso de herramientas computacionales básicas y avanzadas para procesar, analizar, visualizar y modelar datos ómicos —como datos genómicos, transcriptómicos, proteómicos y metabolómicos— utilizando el lenguaje de programación Python. Está diseñado principalmente para estudiantes de posgrado, técnicos, postdoctorantes e investigadores que desean incorporar Python en su flujo de trabajo cotidiano para el análisis de datos globales y de alta dimensión. El diplomado tiene una orientación práctica en un 70%, complementado con un 30% teórico que brinda los fundamentos necesarios para la correcta interpretación de los resultados. Los casos de aplicación están enfocados en biología, medicina, biotecnología y bioinformática. Se utilizará la distribución Anaconda. No se requieren conocimientos previos en programación, ya que el curso comienza desde lo esencial. Este diplomado busca que los participantes se familiaricen y dominen herramientas modernas de análisis reproducible, escalable y automatizable para abordar con confianza datos de alta complejidad en investigación biomédica y ciencias de la vida.
- Profesor: Eliel Ruiz

Aprende a evaluar propiedades ADMET (absorción, distribución, metabolismo, excreción y toxicidad) mediante herramientas de quimioinformática aplicadas al diseño y selección de compuestos bioactivos. Conoce fundamentos de farmacocinética, bases de datos (como PubChem o DrugBank), y plataformas como SwissADME o ADMETsar. Ideal para estudiantes, investigadores y profesionales del área farmacéutica y biomédica.
- Profesor: Andres Alvarado Ssalazar
- Profesor: Juan Andres

l Diplomado está dirigido a estudiantes, investigadores y profesionales del sector empresarial que necesitan aplicar métodos estadísticos sólidos en sus proyectos. Este programa asincrónico aborda temas clave como recolección, organización e interpretación de datos, formulación de hipótesis y aplicación de pruebas estadísticas, todo utilizando el entorno RStudio mediante ejemplos prácticos aplicables a investigaciones reales y análisis de datos empresariales. Se recomienda contar con conocimientos básicos en estadística y un manejo funcional de computadora, incluyendo la instalación de programas, uso de hojas de cálculo, navegación por carpetas, comprensión básica de archivos de texto y ejecución de comandos en software especializado. Al finalizar, los participantes estarán capacitados para aplicar herramientas estadísticas con precisión en contextos académicos, clínicos, sociales, industriales o empresariales, incluyendo análisis de datos en áreas como marketing, producción, control de calidad, biotecnología, y salud.
- Profesor: Roly Ramos
Curso enfocado en el diseño de fármacos asistido por inteligencia artificial. Cubre programación en Python, machine learning, quimioinformática, modelos QSAR y optimización molecular. Ideal para quienes buscan aplicar herramientas computacionales modernas al desarrollo farmacéutico.
- Profesor: ERICK CORREA PADILLA
En este curso el alumno aprenderá conceptos de diseño y tamizaje de fármacos usando quimioinformatica (ADMET), se revisan conceptos de química orgánica básica para ver grupos funcionales, interacciones no covalentes, campos de fuerza y se hará ejercicio para preparar archivos y correr simulación de docking con autodock y hacer análisis de resultados.
En este diplomado el alumno aprenderá conceptos de estructura de proteínas de 1D-4D, el alumnos manejara conceptos de interacciones ligando-proteína no covalentes, aprenderá a realizar estudios de acoplamiento molecular así como estudios de dinámica molecular de proteínas con ligando, en un entorno de membrana, etc. Es un diplomado de bioinformática estructural teórico-práctico desarrollado de la mano de un profesor-investigador experto con publicaciones científicas Internacionales.
- Profesor: Jorge Luis Rosas Trigueros
En este diplomado el alumno aprenderá conceptos de estructura de proteínas de 1D-4D, el alumnos manejara conceptos de interacciones ligando-proteína no covalentes, aprenderá a realizar estudios de acoplamiento molecular así como estudios de dinámica molecular de proteínas con ligando, en un entorno de membrana, etc. Es un diplomado de bioinformática estructural teórico-práctico desarrollado de la mano de un profesor-investigador experto con publicaciones científicas Internacionales.
- Profesor: Gema Ramírez Salinas
En este diplomado el alumno aprenderá conceptos de estructura de proteínas de 1D-4D, el alumnos manejara conceptos de interacciones ligando-proteína no covalentes, aprenderá a realizar estudios de acoplamiento molecular así como estudios de dinámica molecular de proteínas con ligando, en un entorno de membrana, etc. Es un diplomado de bioinformática estructural teórico-práctico desarrollado de la mano de un profesor-investigador experto con publicaciones científicas Internacionales.
- Profesor: Jorge Luis Rosas Trigueros
En este diplomado el alumno aprenderá conceptos de estructura de proteínas de 1D-4D, el alumnos manejara conceptos de interacciones ligando-proteína no covalentes, aprenderá a realizar estudios de acoplamiento molecular así como estudios de dinámica molecular de proteínas con ligando, en un entorno de membrana, etc. Es un diplomado de bioinformática estructural teórico-práctico desarrollado de la mano de un profesor-investigador experto con publicaciones científicas Internacionales.
- Profesor: Gema Ramírez Salinas
En este diplomado el alumno aprenderá conceptos de estructura de proteínas de 1D-4D, el alumnos manejara conceptos de interacciones ligando-proteína no covalentes, aprenderá a realizar estudios de acoplamiento molecular así como estudios de dinámica molecular de proteínas con ligando, en un entorno de membrana, etc. Es un diplomado de bioinformática estructural teórico-práctico desarrollado de la mano de un profesor-investigador experto con publicaciones científicas Internacionales.
- Profesor: Gema Ramírez Salinas
En este diplomado el alumno aprenderá conceptos de estructura de proteínas de 1D-4D, el alumnos manejara conceptos de interacciones ligando-proteína no covalentes, aprenderá a realizar estudios de acoplamiento molecular así como estudios de dinámica molecular de proteínas con ligando, en un entorno de membrana, etc. Es un diplomado de bioinformática estructural teórico-práctico desarrollado de la mano de un profesor-investigador experto con publicaciones científicas Internacionales.
- Profesor: Gema Ramírez Salinas

Proporcionar los fundamentos teóricos y prácticos para evaluar la capacidad antioxidante de compuestos mediante las técnicas de DPPH, ABTS y FRAP, orientado a estudiantes y profesionales en ciencias de la salud, química y áreas afines.
- Profesor: ASTRID RIVERA ANTONIO
En este diplomado se verá de forma integral la bioestadística aplicado al campo farmacéutica y de ciencias de la salud, desde estructura de un proyecto experimental con tamaño de la muestra, grupos problema, testigos, etc. Además aprenderás a organizar la información (datos) para poder comparar entre las poblaciones. Aprenderás a decidir, de acuerdo al diseño del experimento y tamaño de la muestra, el tipo de análisis estadístico que se aplicará. Este diplomado es teórico-práctico, con ejercicios reales. Se usara plataforma virtual www.rcampus.com para trabajar de forma sincrónica, sin embargo, si los alumnos lo consideran, se pueden hacer sesiones sincrónicas con el profesor para aclarar dudas. Todos los ejercicios se realizaran usando los programas: EXCEL, SPSS y graphpad en versiones de prueba académicas.
En este diplomado se verá de forma integral la bioestadística aplicado al campo farmacéutica y de ciencias de la salud, desde estructura de un proyecto experimental con tamaño de la muestra, grupos problema, testigos, etc. Además aprenderás a organizar la información (datos) para poder comparar entre las poblaciones. Aprenderás a decidir, de acuerdo al diseño del experimento y tamaño de la muestra, el tipo de análisis estadístico que se aplicará. Este diplomado es teórico-práctico, con ejercicios reales. Se usara plataforma virtual www.rcampus.com para trabajar de forma sincrónica, sin embargo, si los alumnos lo consideran, se pueden hacer sesiones sincrónicas con el profesor para aclarar dudas. Todos los ejercicios se realizaran usando los programas: EXCEL, SPSS y graphpad en versiones de prueba académicas.
El Diplomado está diseñado para quienes buscan aprender a diseñar, analizar e interpretar datos en investigaciones biomédicas y farmacéuticas. A lo largo del diplomado se abordarán temas desde el diseño de un protocolo experimental, el cálculo del tamaño de muestra y la organización de bases de datos, hasta la selección adecuada de pruebas estadísticas según el tipo de estudio. El Diplomado es teórico-práctico, con ejercicios reales usando Excel, SPSS y GraphPad (en versiones académicas de prueba), y se imparte de manera virtual asincrónica, con posibilidad de sesiones en vivo para resolver dudas. El temario está estructurado en tres niveles: básico, intermedio y avanzado, e incluye temas como estadística descriptiva, pruebas de normalidad, chi cuadrada, ANOVA, regresión, curvas ROC, modelos lineales, validación de instrumentos y más. Se recomienda tener conocimientos básicos en investigación y manejo de hojas de cálculo. Esta formación es útil para estudiantes de posgrado, investigadores, profesionales del área de la salud y personal de la industria farmacéutica que deseen mejorar sus capacidades en análisis de datos y toma de decisiones basada en evidencia.
- Profesor: Cindy Bandala

En este curso aprenderás a realizar predicciones de fosforilación en proteínas usando herramientas computacionales. Además aprenderás a ver los cambios estructurales que estos cambios postraduccionales inducen, particularmente los efectos sobre el reconocimiento molecular.
- Profesor: Jazziel Velazquez Toledano

En el presente curso aprenderás a realizar un Cribado virtual de moléculas para la selección de una molécula o ligando (Hit) con posible actividad sobre un blanco terapéutico (proteína) usando un software de docking robusto y potente, Autodock Vina. Aprenderás a usar diferentes bases de datos de moléculas de las cuales puedes extraer una gran cantidad de ligandos para su evaluación por computadora y hacer un cribado o filtrado con la finalidad de elegir la mejor o mejores contra un blanco terapéutico. En el curso se da una descripción detallada y se va guiando al estudiante paso a paso desde la selección de las moléculas a evaluar, el blanco molecular así como su preparación, evaluación y selección del mejor ligando. El curso esta diseñado para Windows y Linux.
- Profesor: Yudibeth Sixto López

Química Cuántica IIA
En este curso, exploraremos los principios y las aplicaciones de la química cuántica moderna utilizando como referencia el libro "Modern Quantum Chemistry" de Attila Szabo. Nuestro objetivo principal es comprender la notación básica, las operaciones matemáticas y aproximaciones utilizados en la descripción cuántica de los sistemas moleculares, abordando tanto los fundamentos matemáticos como las herramientas computacionales más relevantes en el campo.
Contenidos del curso:
I. Introducción a las matemáticas:
- Álgebra lineal aplicada a la mecánica cuántica.
- Operadores cuánticos.
- Método variacional.
II. Repaso de las soluciones de la ecuación de Schrödinger:
- Aproximación de Born-Oppenheimer y su relevancia en sistemas moleculares.
III. El problema de la función de onda multielectrónica:
- Descripción de los espines orbitales.
- Principio de exclusión de Pauli.
- Determinantes de Slater.
IV. Operadores y elementos de matriz:
- Introducción a la notación matemática.
- Reglas generales y operadores de espín.
V. Aproximación de Hartree-Fock:
- Ecuaciones y soluciones de Hartree-Fock.
- Ecuaciones de Roothaan y su interpretación.
VI. Conjunto de bases poliatómicas:
- Uso de funciones gaussianas contraídas.
- Conjuntos de bases STO-3G y polarizadas.
VII. Cálculos de capa abierta y cerrada Hartree-Fock:
- Diferencias entre cálculos de capa cerrada y abierta.
Actividades complementarias:
Actividad 0: Instalación de programas y visualizadores
En esta actividad, instalaremos las herramientas computacionales necesarias como GAMESS, Avogadro, Gabedit, Macmolplt y Multiwfn.
Actividad 1: Optimización de geometría y cálculo de frecuencias
Aprenderemos los comandos básicos para realizar optimizaciones de geometría, cálculos de frecuencias y análisis conformacional usando métodos y bases diversas.
Práctica Computacional:
Los estudiantes realizarán cálculos utilizando el programa XTB, GAMESS y ORCA para obtener energías, frecuencias vibracionales, potenciales de ionización, análisis de población y momentos dipolares, visualizando los resultados con Avogadro.
¡Espero que disfruten el curso y que les resulte de gran utilidad en su formación académica y profesional!
- Profesor: Brena Manzanilla
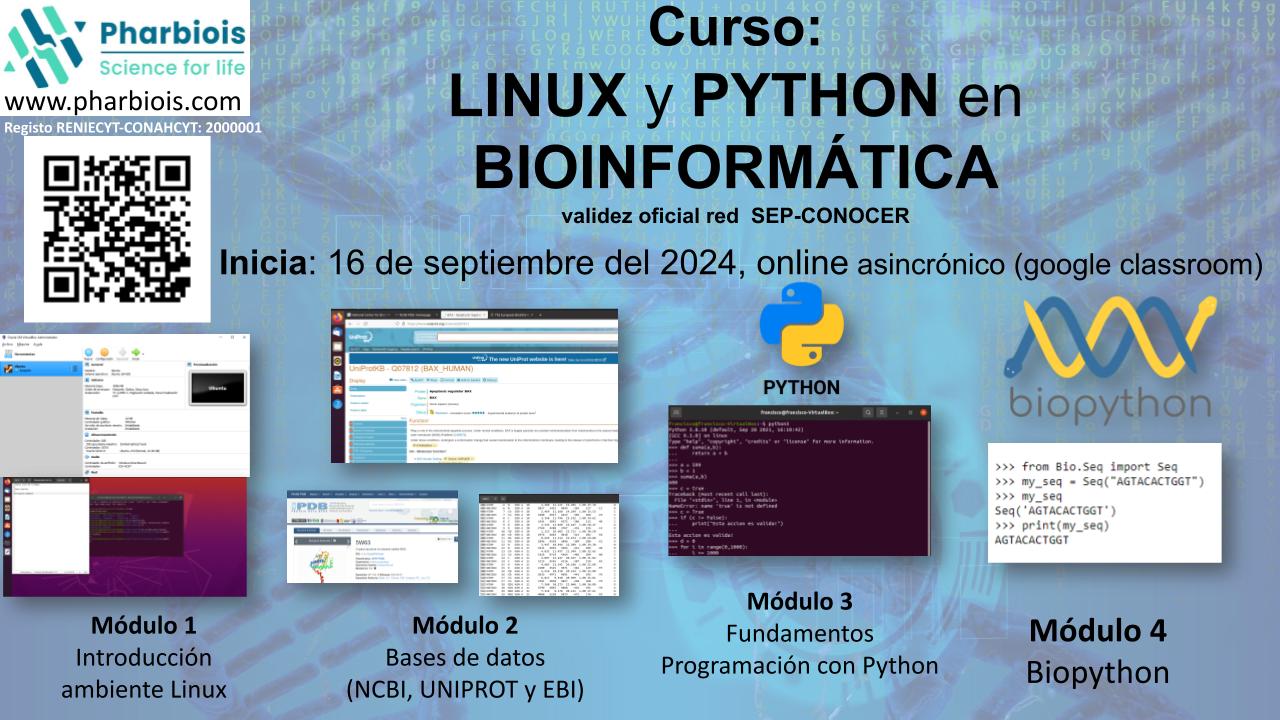
Aprende a manejar el sistema operativo LINUX en tu computadora y/o de forma remota, además en este curso aprenderás PYTHON y BIOPYTHON aplicado al análisis de información biológica con enfoque BIOINFORMÁTICO, QUIMIOINFORMÁTICA, además para programar en R y PERL todo esto de la mano de informáticos expertos en la temática.
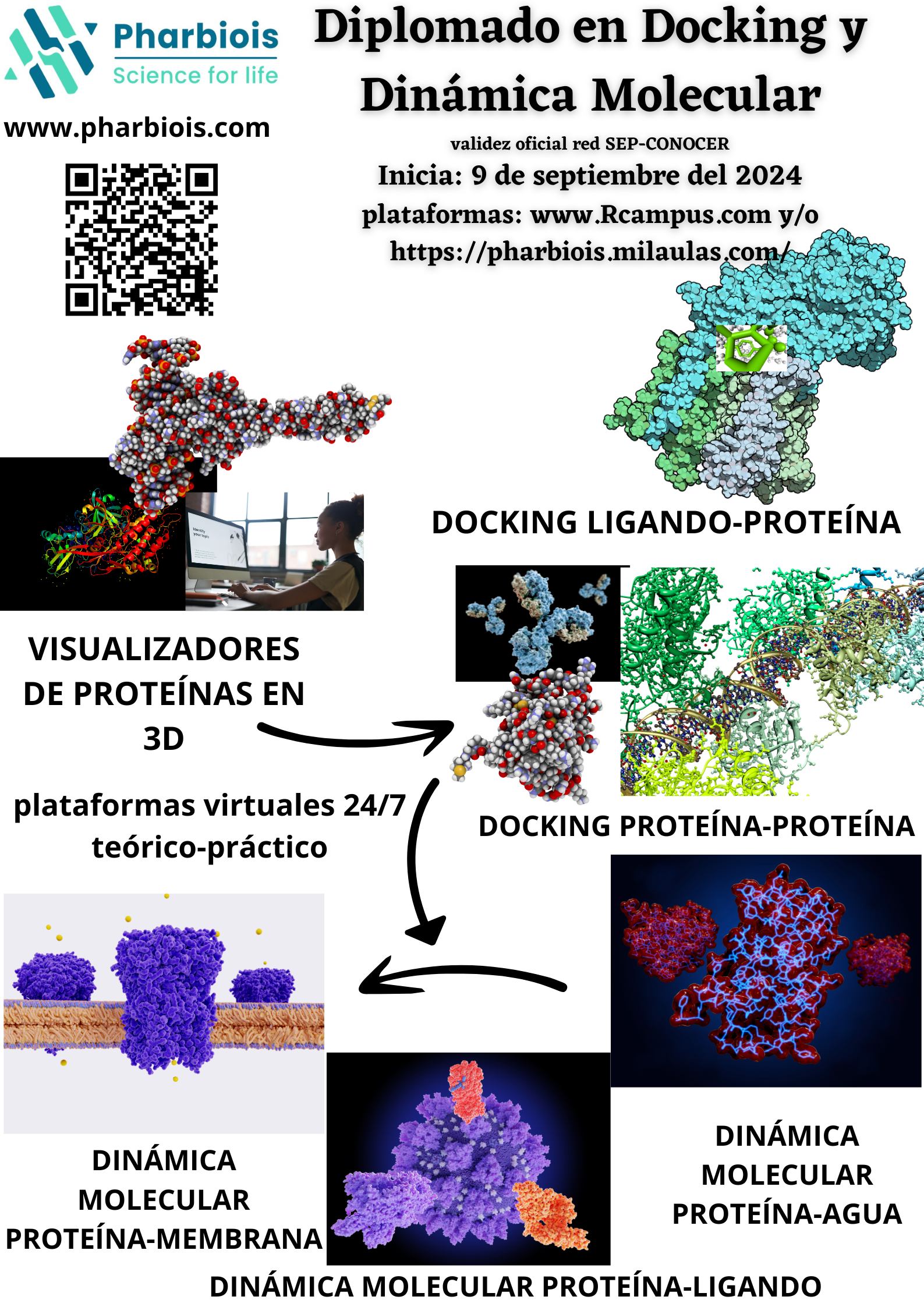
En este diplomado el alumno aprenderá conceptos de estructura de proteínas de 1D-4D, el alumnos manejara conceptos de interacciones ligando-proteína no covalentes, aprenderá a realizar estudios de acoplamiento molecular así como estudios de dinámica molecular de proteínas con ligando, en un entorno de membrana, etc. Es un diplomado de bioinformática estructural teórico-práctico desarrollado de la mano de un profesor-investigador experto con publicaciones científicas Internacionales.
- Profesor: Gema Ramírez Salinas

Este curso está dirigido a quienes deseen profundizar en el acoplamiento molecular incorporando la flexibilidad del receptor con ADFR (AutoDock for Flexible Receptors), sucesor de AutoDock y AutoDock Vina. Aprenderás a preparar archivos, ejecutar simulaciones y analizar resultados usando UCSF Chimera y Avogadro. Se recomienda haber tomado el Curso de Docking Proteína-Ligando con AutoDock, además de tener conocimientos en biología molecular, formatos PDB o FASTA y nociones básicas de bioinformática. Lo aprendido se aplica en el diseño y reposicionamiento de fármacos, investigación biomédica y proyectos farmacéuticos o industriales.
- Profesor: Lenin Dominguez Ramierz

En este curso el alumno aprenderá a diseñar sus primers o iniciadores, además de revisar conceptos sobre los pesos moleculares en geles así como el manejo de herramientas bioinformáticas para diseño y análisis.
- Profesor: Ismael Vasquez Moctezuma

Aprenderás a identificar oportunidades de negocio, evaluar la competencia y comprender mejor el panorama de la propiedad intelectual en la industria farmacéutica. Te guiaremos a través de ejemplos prácticos y casos de estudio, brindándote las herramientas necesarias para tomar decisiones informadas en tu investigación y desarrollo de productos.
- Profesor: Rolando Alberto Rodríguez Fonseca
Este curso permite comprender el comportamiento de átomos y moléculas desde un enfoque matemático, esencial para el uso de herramientas computacionales en química teórica. Se recomienda haber cursado previamente el Curso de Métodos de Estructura Electrónica y tener bases en física y matemáticas. Se abordarán temas como la radiación del cuerpo negro, efecto fotoeléctrico, hipótesis de De Broglie, ecuación de Schrödinger, principio de incertidumbre, operadores y modelos clave como partícula en una caja, oscilador armónico y átomo hidrogenoide. En la práctica computacional se realizará optimización de geometría y cálculo de frecuencias para espectros IR. Al finalizar, el estudiante podrá interpretar modelos cuánticos y realizar simulaciones moleculares básicas.
- Profesor: Brena Manzanilla

Es un curso que te dará conceptos y y práctica sobre estructura primaria, secundaria y terciara de proteínas, aprenderás a buscar secuencias, a usar herramientas bioinformáticas para construir estructuras 3D. Además aprenderás conceptos y manejo de herramientas para realizar evaluación de estructural. La práctica la realizaras tu mismo en tu computadora con acceso remoto a servidores, instalación de herramientas en tu máquina, etc.
- Profesor: Gema Mondragón
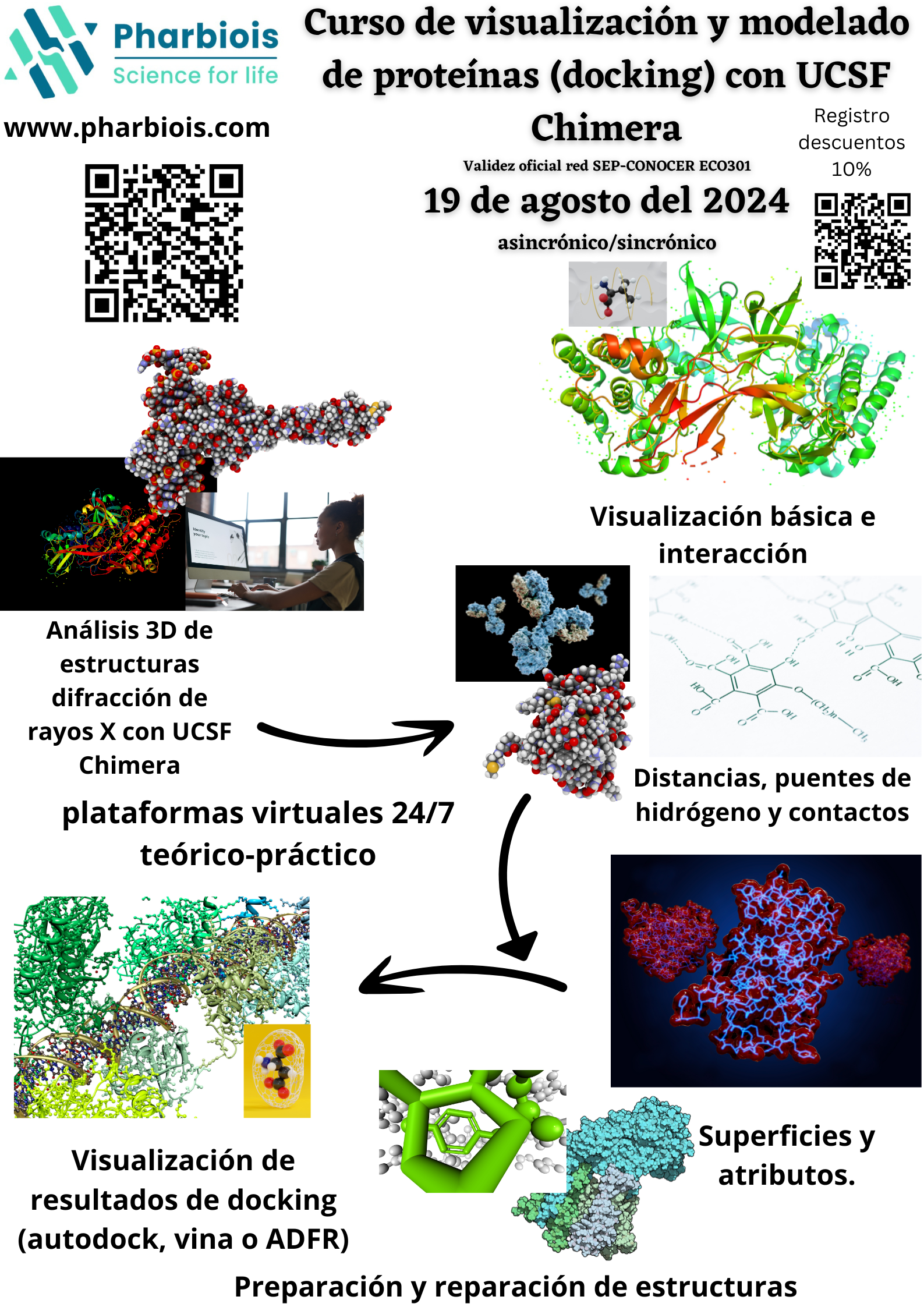
Curso práctico con UCSF Chimera para estudiantes, investigadores y profesionales del sector farmacéutico con conocimientos básicos en biología molecular y formatos PDB/FASTA. Aprenderás a visualizar, analizar y preparar estructuras proteicas desde su secuencia hasta su uso en docking molecular. Aplicarás estos conocimientos en investigación biomédica, modelado estructural, y desarrollo de fármacos en entornos académicos e industriales.
- Profesor: Lenin Dominguez Ramierz

El Curso (módulo 2) está dirigido a estudiantes e investigadores en química, biología, biomedicina y nanotecnología interesados en el diseño racional de fármacos. A lo largo del curso, se aprenderá a analizar propiedades químicas, modelar proteínas y diseñar moléculas bioactivas aplicando principios de QSAR, ADMET, mecánica molecular, mecánica cuántica y dinámica molecular. Se abordarán técnicas de docking ligando-proteína, interacción proteína-proteína y el uso de dendrímeros como nanoacarreadores. El curso cierra con talleres prácticos utilizando herramientas como VMD y NAMD. Para un mejor aprovechamiento, se recomienda contar con conocimientos previos en biología molecular, química orgánica, estructura de proteínas y manejo básico de computadora. Este aprendizaje tiene amplias aplicaciones futuras en investigación científica, desarrollo farmacéutico, optimización de compuestos candidatos y colaboración en proyectos de nanotecnología y bioinformática estructural.
- Profesor: José Correa Basurto

La mayoría de estudiantes e investigadores presentan dificultades al momento de elegir y aplicar el método estadístico para su investigación, de manera que los resultados obtenidos no responden a los objetivos establecidos. El presente diplomado desarrolla de forma asincrónica la recolección, organización, elaboración de hipótesis y pruebas estadísticas con el programa Rstudio e interpretación adecuada. El desarrollo del curso se da a través de ejemplos directos en posibles investigaciones o similares a realizar
- Profesor: Roly Ramos

La Quimioinformática es una de las disciplinas que se ha convertido en un pilar durante el desarrollo y diseño de nuevos fármacos y, por lo tanto, indispensable en el menester de la Química Farmacéutica. Esta área del conocimiento permite resolver problemas en el manejo y presentación de información en química mediante la integración de diferentes conceptos, técnicas y métodos computacionales.
- Profesor: Brayan Raziel Cedillo Gonzalez
- Profesor: Jose Medina-Franco
- Profesor: Diana Prado
- Profesor: Fernanda Saldivar

Las aplicaciones de la biotecnología moderna se fundamentan en gran medida en conocer las bases de las ciencias ómicas, tales como genómica, transcriptómica, y proteómica. Todo ello ha sido posible por la comprensión y optimización de dichos conocimientos, de los cuales se puede destacar el estudio de la genética de los plásmidos.
- Profesor: Javier David Uzcategui Gonzalez

En este curso de minería de datos en Rstudio aprenderás a instalar y manejar los comandos básicos de programación en R, no te pierdas este interesante e importante curso, muy útil hoy en día para manejo masivo de datos aplicado a la minería de datos.
- Profesor: Roly Ramos

Los estudios de relaciones Cuantitativas Estructura - Actividad (QSAR) tienen como objetivo la búsqueda de correlaciones entre descriptores moleculares y actividades químicas o biológicas para la predicción de las mismas en compuestos desconocidos. Un modelo validado supera el vínculo casuístico entre descriptores y actividades, y se convierte en una herramienta predictiva.
Objetivos:
- Conocer los fundamentos técnicos y científicos de un estudio QSAR.
- Conocer aspectos regulatorios de las metodologías QSAR
- Profesor: Bruno Hernández
- Profesor: Abraham Madariaga Mazón
- Profesor: Karina Martinez

Introducción
Este diplomado está diseñado para proporcionar los conocimientos básicos necesarios para realizar cálculos computacionales en química. El objetivo es capacitar a los estudiantes para que puedan obtener resultados confiables en la modelización y simulación de sistemas químicos, basándose en una sólida comprensión teórica y en la aplicación práctica de software especializado.
---
Objetivos del Diplomado
Comprender la teoría fundamental: Proveer una base teórica sobre el funcionamiento de los programas de cálculo de estructura electrónica.
Aplicación práctica: Realizar cálculos computacionales usando software libre para el aprendizaje.
Obtención de resultados confiables: Enseñar a interpretar los resultados obtenidos y su relevancia en la química computacional.
Propiedades y reactividad química: Calcular propiedades químicas, reactividad y termoquímica de diferentes sistemas químicos.
---
Contenido del Diplomado
1. Fundamentos Teóricos
- Introducción a la Química Computacional.
- Principios de la Mecánica Cuántica y su aplicación en cálculos computacionales.
- Métodos de estructura electrónica: Teoría del Funcional de la Densidad (DFT), Métodos Ab Initio, etc.
- Conceptos básicos de reactividad y propiedades químicas.
2. Aplicación Práctica
- Introducción al uso de software libre para cálculos computacionales.
- Configuración y ejecución de simulaciones.
- Análisis e interpretación de resultados.
- Casos de estudio: propiedades y reactividad de moléculas sencillas y complejas.
---
Metodología
- Clases teóricas: Explicaciones detalladas de los conceptos fundamentales.
- Ejercicios y casos prácticos: Aplicación de los conocimientos adquiridos en problemas reales.
---
Software Utilizado Libre (se ajustará a las necesidades)
-GAMESS
- ORCA
- NWChem
- Avogadro
---
Requisitos
- Conocimientos básicos en química y física.
- Familiaridad con conceptos de matemáticas y programación es deseable, pero no indispensable.
---
Duración y Modalidad
- Duración: 3 meses
- Modalidad: Online, con sesiones en vivo (si así se desea) y materiales disponibles para autoaprendizaje.
- Profesor: Brena Manzanilla

La mayoría de estudiantes e investigadores presentan dificultades al momento de elegir y aplicar el método estadístico para su investigación, de manera que los resultados obtenidos no responden a los objetivos establecidos. El presente curso asincrónico pretende guiar en la elaboración de hipótesis y objetivos a partir de métodos multivariados así como su manejo estadístico con el programa Rstudio e interpretación adecuada. El desarrollo del curso se da a través de ejemplos directos en posibles investigaciones o similares a realizar.
- Profesor: Roly Ramos

La aplicación y la pertinencia de un modelo QSAR se basa en el conjunto de elementos empleados para la obtención de la correlación y en los métodos estadísticos para evaluarlo. La correcta selección de los conjuntos moleculares para la generación del modelo QSAR resulta de suma importancia en la validación de este, ya que debe guardar relación con el espacio químico bajo estudio. Esta serie de cursos tienen como objetivo brindar las herramientas necesarias para determinar la aplicación, la pertinencia y calidad de un estudio QSAR.
Objetivos:
- Familiarizar al participante con los principios de la OECD para el desarrollo de modelos QSAR
- Conocer los principales programas computacionales utilizados en el desarrollo de modelos (Q)SAR.
- Profesor: Bruno Hernández
- Profesor: Abraham Madariaga Mazón
- Profesor: Karina Martinez

Este curso ofrece fundamentos teórico-prácticos en bioinformática estructural aplicada, enfocados en la predicción y análisis de interacciones proteína-proteína mediante docking molecular. Aprenderás a utilizar servidores como HDOCK, ClusPro, HADDOCK, entre otros, para modelar y evaluar interfaces proteicas con aplicaciones en procesos biológicos y el diseño de nuevas estrategias terapéuticas. Requisitos: conocimientos básicos de química general, bioquímica de proteínas, y recomendable haber cursado previamente el "Curso de Docking Proteína-Ligando con Autodock". Esta formación es altamente útil en investigación académica, desarrollo de fármacos y el sector industrial biotecnológico.
- Profesor: Alberto Guillen

Curso (módulo) dirigido a estudiantes, investigadores y empresas farmacéuticas con conocimientos básicos en biología molecular, estructura de proteínas y manejo de computadora. Aprenderás a ejecutar simulaciones de dinámica molecular con NAMD, VMD y CARMA, interpretarás métricas como RMSD y RMSF. Aplicarás estos conocimientos en investigación biomédica, diseño racional de fármacos y biotecnología estructural
- Profesor: José Correa Basurto
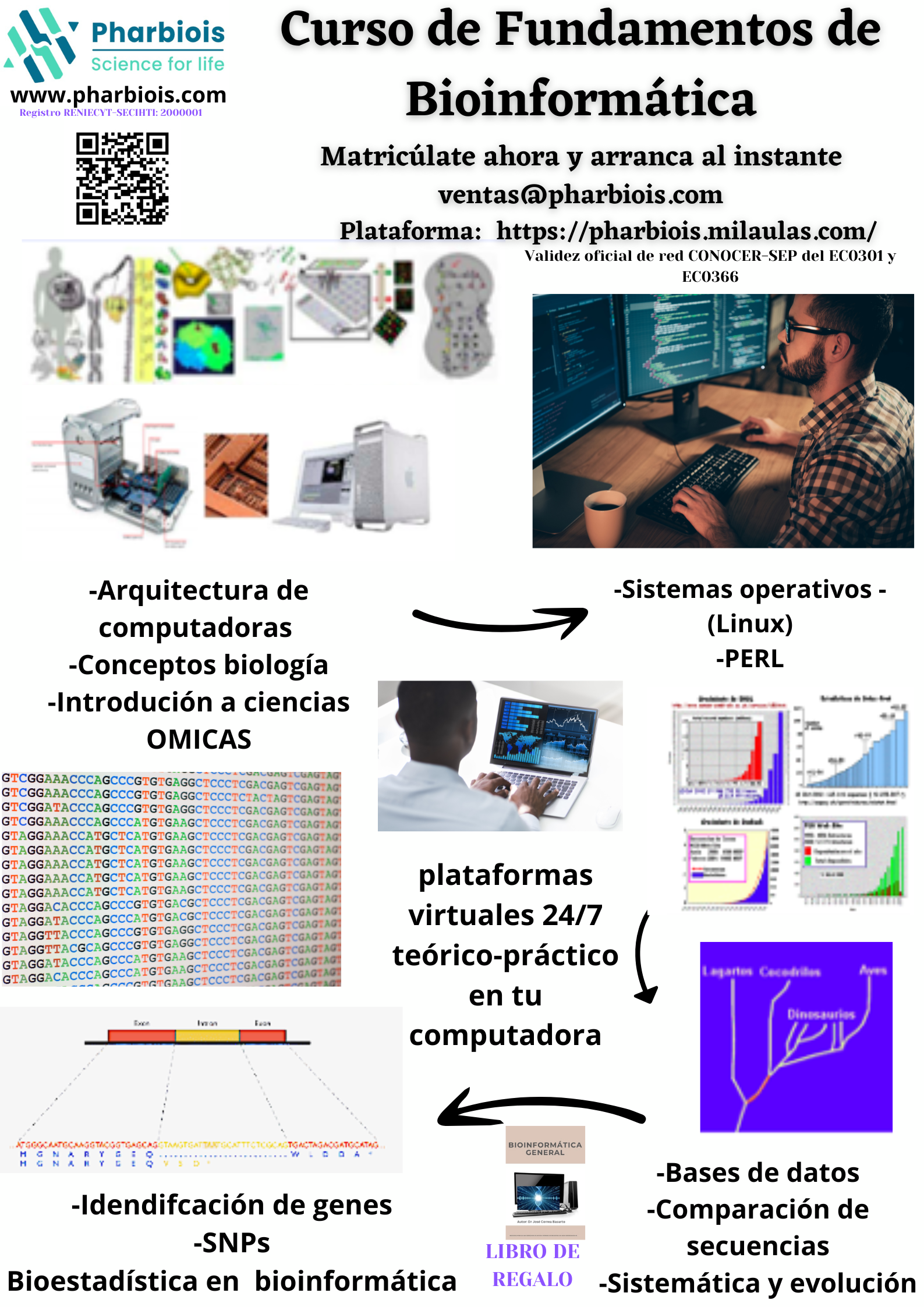
Este curso (módulo 1) brinda una formación integral desde las bases computacionales hasta el análisis avanzado de datos biológicos, ideal para estudiantes e investigadores en áreas biológicas, biomédicas y biotecnológicas. Aprenderás el manejo básico de sistemas operativos Linux, fundamentos de programación en Perl, análisis de secuencias, herramientas OMICS, quimioinformática y bioestadística aplicada. Se recomienda contar con conocimientos básicos en biología molecular y manejo funcional de computadora (uso de archivos, navegación de carpetas y comprensión básica de comandos). La modalidad es práctica, flexible y enfocada en problemas reales. Al finalizar, podrás desarrollar un proyecto de alineamiento y construcción de árboles filogenéticos con proteínas reales. Este aprendizaje te preparará para aplicar la bioinformática en investigación académica, desarrollo biotecnológico, salud, farmacogenómica y exploración de bases de datos moleculares.
- Profesor: José Correa Basurto
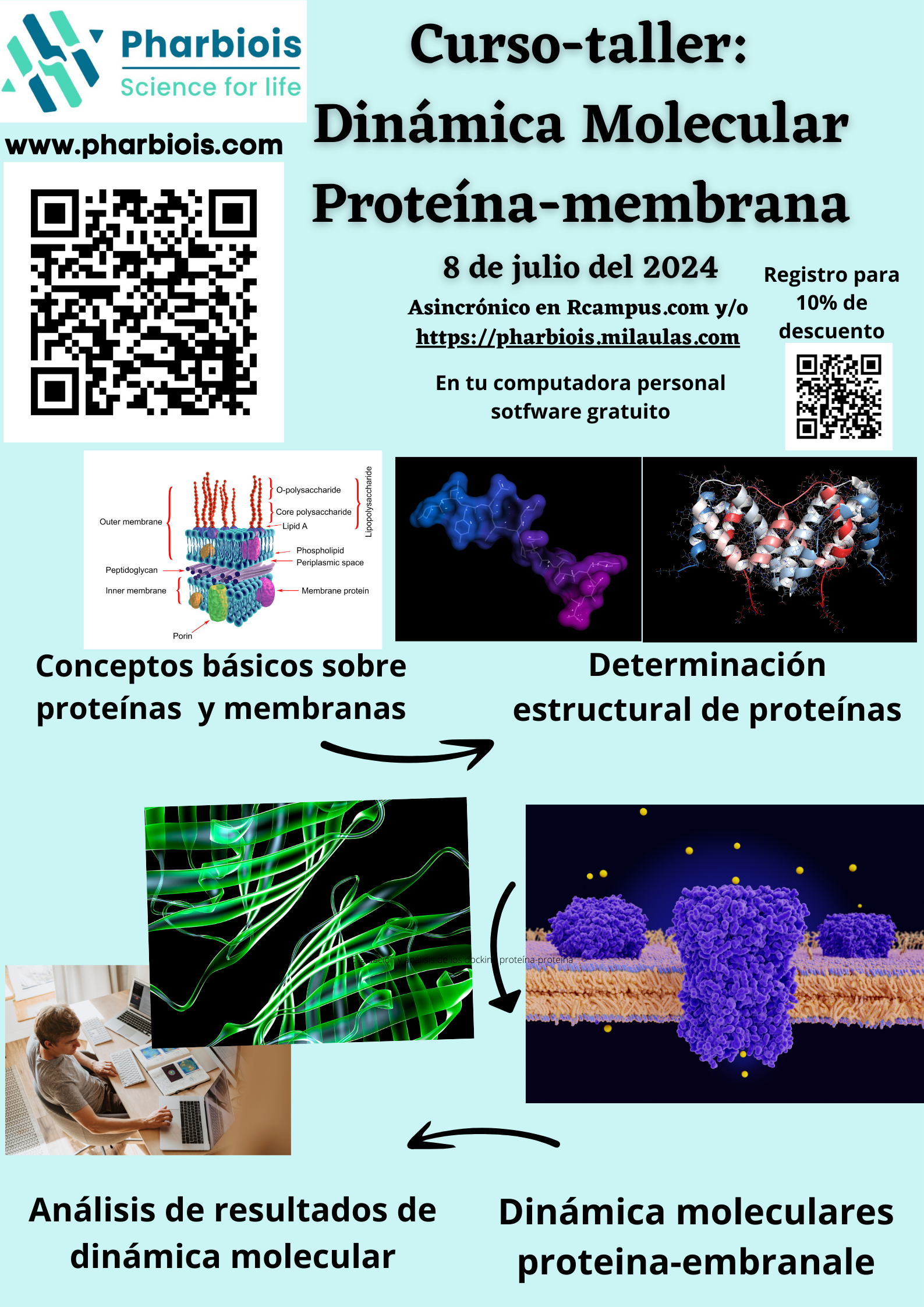
acerca del curso
Este curso te permitirá aprender conceptos sobre estructura de proteínas, además de la importancia de las proteínas en un entorno lipídico para todas aquellas proteínas transmembranales, harás ejercicios para insertar la proteína en membrana y correr una dinámica de un sistema con estas características además de analizar tus resultados cómo RMSD, Rg, RMSF, interacciones no covalentes proteína, cambios conformacionales, etc. Para realizar este curso se requiere tener conocimientos en estructura terciaria de proteínas, conocimientos en membranas lipídicas, manejo básico de linux en modo texto.
- Profesor: Gema Ramírez Salinas

En este curso de R aprenderás a instalar y manejar los comandos básicos de programación en R, no te pierdas este interesante e importante curso, muy útil hoy en día para manejo masivo de datos, es una herramienta gratuita de programación con gran uso potencial en bioestadística, bioinformática de secuencias, bioinformática estructural, datos de omics (metabolómica, lipidómica, proteómica etc), big data etc.
- Profesor: Roly Ramos

Este curso te permitirá aprender conceptos sobre estructura de proteínas, ligandos, campos de fuerza, e interacciones no covalentes proteína-ligando, cambios conformacionales, preparar tu sistema para correr dinámica molecular proteína-ligando, correr tu simulación, analizar tus resultados cómo rmsd, Rg, rmsf, interacciones no covalentes proteína-ligando, calculó de energía, etc.
- Profesor: Gema Ramírez Salinas

Métodos de Estructura Electrónica
Descripción del curso:
Este curso ofrece una comprensión profunda de la estructura electrónica tanto a nivel atómico como molecular. Los estudiantes aprenderán los fundamentos de los métodos aproximados utilizados en química computacional y cómo aplicarlos para realizar cálculos computacionales. Al finalizar el curso, los participantes serán capaces de:
- Determinar geometrías óptimas de moléculas y complejos atómicos.
- Calcular energías electrónicas con precisión.
- Evaluar y predecir propiedades moleculares esenciales.
Objetivos del curso:
-
Comprender la estructura electrónica:
- Fundamentos de la teoría de la estructura electrónica.
- Diferenciación entre estructuras atómicas y moleculares.
-
Métodos aproximados en química computacional:
- Introducción a los métodos de Hartree-Fock y DFT (Teoría del Funcional de la Densidad).
-
Cálculos computacionales:
- Uso de software especializado para realizar cálculos de química computacional gratuito.
- Determinación de geometrías moleculares óptimas.
- Cálculo de energías electrónicas.
-
Propiedades moleculares:
- Predicción y análisis de propiedades físicas y químicas.
- Interpretación de resultados computacionales para aplicaciones prácticas.
- Profesor: Brena Manzanilla
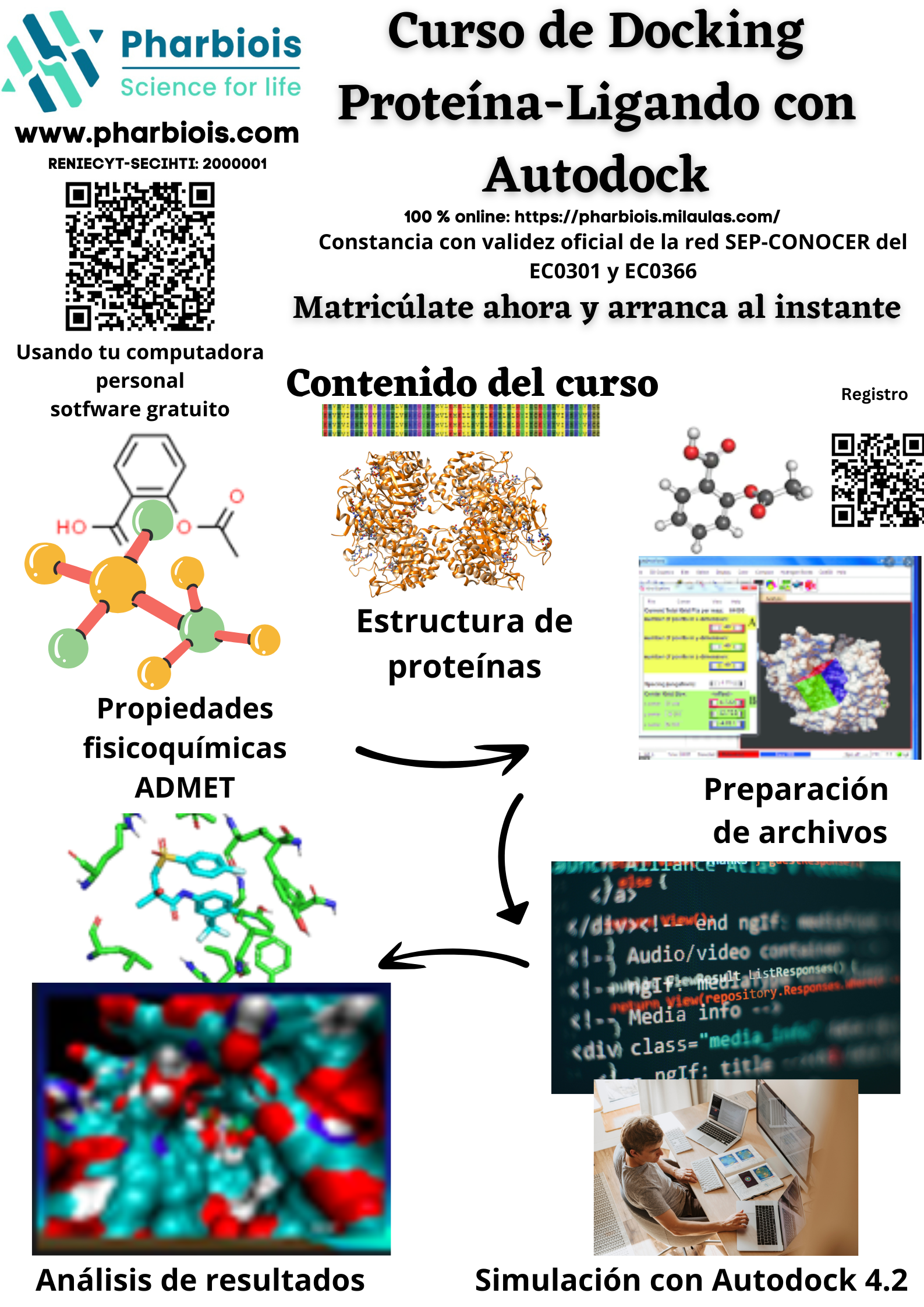
El Curso (módulo 4) está dirigido a estudiantes e investigadores de áreas como química, biología, bioquímica, farmacología o afines, que cuenten con conocimientos básicos en química orgánica, biología molecular y manejo de computadora. En este curso aprenderás los fundamentos clave de la química orgánica aplicada al diseño racional de fármacos, incluyendo el análisis de grupos funcionales, propiedades ADMET, interacciones no covalentes y campos de fuerza. Utilizando herramientas de quimioinformática, realizarás el diseño y tamizaje de compuestos, así como la preparación de ligandos y proteínas blanco. Se te guiará paso a paso en la instalación y uso de AutoDock y AutoDockTools (ADT), la ejecución de simulaciones de acoplamiento molecular (docking), el análisis de resultados y la validación estructural de los complejos generados. El curso culmina con un proyecto práctico de docking completo, aplicado a casos reales. Esta formación práctica y aplicada te permitirá desarrollar competencias esenciales para la investigación en diseño de fármacos asistido por computadora, así como para aplicaciones en farmacología computacional, innovación en biotecnología y proyectos de desarrollo farmacéutico académico o empresarial.
- Profesor: José Correa Basurto
addactividades
- Profesor: pharbiois science for life

El curso (módulo 3) está diseñado para estudiantes e investigadores en ciencias biomédicas, biotecnológicas o afines que cuenten con conocimientos previos en inmunología básica, biología molecular y manejo general de computadora. Durante el curso, se abordarán los fundamentos esenciales de la inmunología enfocados en el desarrollo racional de vacunas basadas en epítopes, tanto para la inmunidad humoral como celular. Aprenderás a identificar y predecir epítopes lineales y conformacionales, así como a analizar su reconocimiento por moléculas del MHC-I y MHC-II. Además, realizarás modelado estructural de proteínas y estudios de acoplamiento molecular, utilizando como modelo la proteína Spike del virus SARS-CoV-2. También se aplicarán estrategias de bioingeniería para el diseño de nanovacunas, acoplando epítopes a nanopartículas como los dendrímeros PAMAM-G4. Este curso teórico-práctico, impartido en modalidad flexible y aplicada, proporciona una base sólida para el desarrollo de proyectos en inmunología computacional, investigación biomédica, diseño de vacunas y terapias basadas en péptidos y nanotecnología.
- Profesor: José Correa Basurto